物种的系统发育树可以展现地球上现存物种间的进化关系。微观尺度上,一个生物个体所有细胞的亲缘关系可以用细胞谱系树来描述。细胞谱系对于理解多细胞生物的胚胎发育、细胞类型的分化、器官的维持与衰老等生理病理过程具有重要意义,是分子进化生物学与发育生物学的交叉前沿。1983年,John Sulston等人利用显微镜观察并记录了秀丽隐杆线虫胚胎发育过程671细胞的分裂历史,首次获得一个多细胞生物完整的细胞谱系树【1】。随后放射性元素、酶和荧光标记等方法也逐渐应用于细胞亲缘关系的追踪,但效果欠佳。近年,体细胞的突变信息被用于细胞谱系追踪【2-4】。为了解决本底突变率较低的问题,人们着力于发展基于DNA条形码(barcode)的技术,借助Cre-LoxP、CRISPR-Cas9等高效的基因编辑工具,对特定的DNA条形码区域进行编辑【5,6】,通过记录伴随细胞分裂过程产生的突变来还原细胞谱系。然而,目前的条形码技术平均每个条形码只携带3-5个突变,远不足以描述复杂多细胞生物胚胎发育的全貌。
2021年12月2日,中山大学生命科学学院贺雄雷教授团队在Nature Methods上发表了题为“Mapping single-cell-resolution cell phylogeny reveals cell population dynamics during organ development”的研究论文。文章介绍了一种基于单碱基突变的细胞谱系追踪系统SMALT(substitution mutation aided lineage tracing system)。该系统具有极大的标记空间,含超过1000个潜在突变位点,单个条形码平均可记录超过20个突变,适用于对复杂多细胞生物进行谱系追踪。
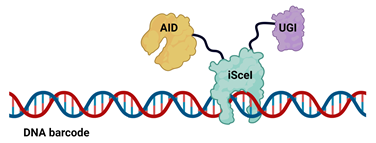
SMALT系统的工作原理是利用脱氨酶(AID)通过iSceI蛋白功能域靶向到DNA条形码序列,将序列上的C碱基通过脱氨反应变成U碱基;在DNA复制时,U碱基会与A碱基配对,从而产生C>T突变(图1)。研究人员在自主搭建的酵母筛选平台中对SMALT系统的各个元件进行了优化,继而通过构建SMALT系统转基因果蝇品系,首次实现在单细胞水平上对复杂多细胞生物的胚胎发育过程中的细胞谱系关系进行可靠描述(图2)。为了解细胞谱系对个体发育与表型稳定性的贡献提供了新的研究思路。
图1 SMALT系统示意图
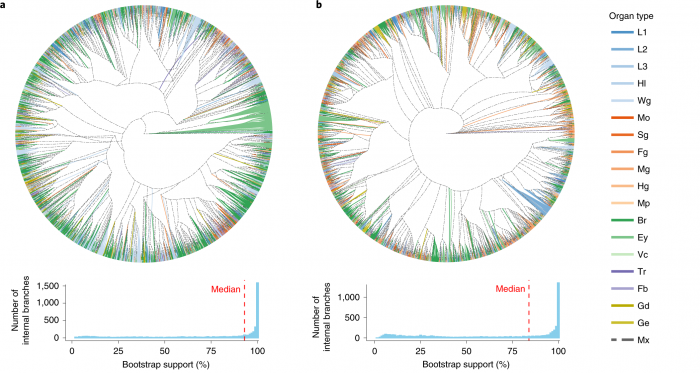
图2 在两个果蝇个体中构建高分辨细胞谱系树
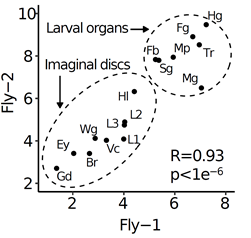
复杂多细胞生物具有海量的细胞数目,对所有细胞进行捕获是不现实的,这意味着重构得到的细胞谱系树只是个体细胞谱系树的子集。对同一个体进行重复抽样可能得到不同的细胞群体。因此,对于复杂多细胞生物细胞谱系的研究不能停留在对谱系树的构建与直观的描述上,应该尝试利用数学模型对谱系树进行量化,推断其反映的细胞群体历史,提取更深层的生物学特征。为此,研究人员借助群体遗传学的溯祖理论,开发了利用细胞谱系树还原具有分裂活性的祖先细胞群体数量的分析方法(an instantaneous coalescence estimation of parental cells with active division, ICPD)。该方法不依赖于群体结构,可从细胞谱系树推断出各器官发育过程的群体历史特征。他们发现,不同器官具有独特的群体历史特征,并且该特征在不同个体具有高度一致性(图3)。相较于成虫盘,幼虫器官具有更复杂的起源历史【7,8】。
图3 果蝇幼虫各器官的分歧时间
尽管每个细胞个体的生长与分裂速度各不相同,但器官大小与形状在群体中却有惊人的重复性。通过ICPD分析提取的群体历史特征,研究人员对各器官的新生细胞数进行了推断,发现各器官中分裂产生的细胞总数与器官的真实细胞数量显著相关,对其方差解释度达到74.5%,揭示了细胞群体的分裂活性对器官大小的稳定性的贡献,进一步说明细胞谱系树中包含了复杂多细胞生物胚胎发育中的底层逻辑(图4)。
图4 累计细胞数量与真实细胞数量间具有一致性
该研究开发的细胞谱系追踪系统以及基于细胞谱系树的群体遗传学分析方法,为还原复杂多细胞生物胚胎发育的过程提供了新的技术手段与研究方法,结合单细胞多组学研究方法,有望对哺乳动物器官发育的鲁棒性、细胞命运转换机制等发育生物学的核心问题作出定量解析。
Reference
1. Sulston, J. E., Schierenberg, E., White, J. G. & Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Dev Biol 100, 64-119, doi:10.1016/0012-1606(83)90201-4 (1983).
2. Lee-Six, H. et al. Population dynamics of normal human blood inferred from somatic mutations. Nature 561, 473-478, doi:10.1038/s41586-018-0497-0 (2018).
3. Frumkin, D., Wasserstrom, A., Kaplan, S., Feige, U. & Shapiro, E. Genomic variability within an organism exposes its cell lineage tree. PLOS Comput. Biol. 1, 382-394, doi:10.1371/journal.pcbi.0010050 (2005).
4. Salipante, S. J. & Horwitz, M. S. Phylogenetic fate mapping. Proc. Natl. Acad. Sci. U.S.A. 103, 5448-5453, doi:10.1073/pnas.0601265103 (2006).
5. Pei, W. et al. Polylox barcoding reveals haematopoietic stem cell fates realized in vivo. Nature 548, 456-460, doi:10.1038/nature23653 (2017).
6. McKenna, A. et al. Whole-organism lineage tracing by combinatorial and cumulative genome editing. Science 353, aaf7907, doi:10.1126/science.aaf7907 (2016).
7. Buchon, N. et al. Morphological and molecular characterization of adult midgut compartmentalization in Drosophila. Cell Rep 3, 1725-1738, doi:10.1016/j.celrep.2013.04.001 (2013).
8. Miguel-Aliaga, I., Jasper, H. & Lemaitre, B. Anatomy and Physiology of the Digestive Tract of Drosophila melanogaster. Genetics 210, 357-396, doi:10.1534/genetics.118.300224 (2018).